央广网上海12月16日消息(采访人员杨静 通讯员潘少军 贾龙豪)复旦大学类脑研究院青年研究员路易斯·佩德罗·科埃略(Luis Pedro Coelho)、教授赵兴明、名誉教授皮尔·伯克(Peer Bork)与来自德国、西班牙、美国、英国等多国科学家合作研究 , 基于全球微生物组(global microbiome)的概念 , 将地球上不同栖息地的微生物作为统一系统 , 运用人工智能技术对1.3万个公开宏基因组样本进行挖掘 , 构建了迄今为止最全面的全球微生物基因目录(GMGC, Global Microbial Gene Catalog) , 为全球微生物组研究迈出了重要一步 。 该研究同时发现 , 大多数基因具有栖息地特异性 , 跨越多栖息地的基因主要富集在抗生素耐药性基因和移动遗传元件 。
北京时间2021年12月16日凌晨 , 相关研究成果《原核生物基因的生物地理学研究》(“Towards the biogeography of prokaryotic genes”)以长文(Article)形式发表于《自然》(Nature)主刊 。 科埃略是论文的第一作者和共同通讯作者 。 该研究得到了欧盟“地平线2020”创新计划、国家重点研发计划、国家自然科学基金、上海市“脑与类脑智能基础转化应用研究”市级科技重大专项等项目的资助 。
构建迄今为止最全面的全球微生物基因目录
基因目录对于描述微生物群落的物种组成和功能特性具有重要意义 。 自2010年欧洲分子生物学实验室(EMBL)和华大基因构建首个人类肠道微生物基因目录以来 , 新兴的微生物基因目录为研究人类生理学和疾病提供了重要线索 。
全球微生物基因目录涵盖了肠道、口腔、皮肤、海洋、土壤等14微生物的主要栖息地 , 收集了13174个公开可用的高质量宏基因组和84029个高质量的基因组 , 得到了包含3.03亿个物种级的基因(95%的核酸一致性聚类) , 构建了迄今为止最全面的全球微生物基因目录 , 将为地球生态研究和人类健康研究提供重要贡献 。
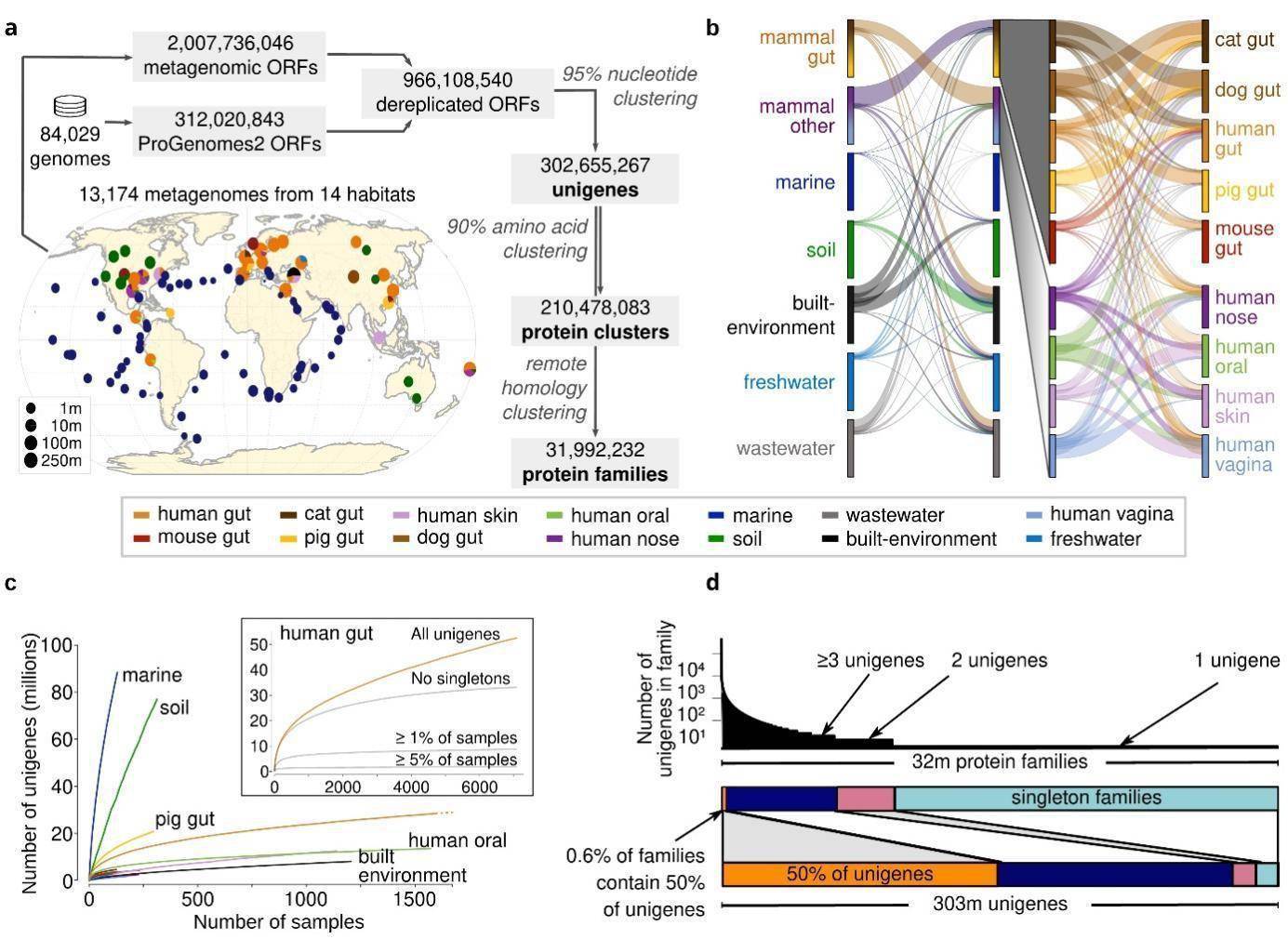
文章图片
从1.3万个宏基因组样本和近10万个细菌基因组中构建的包含3亿个原核生物基因的全球微生物基因目录
揭示微生物基因与栖息环境的重要关联
物种水平的单基因簇(unigene)可能代表着来自多种栖息地的基因(multi-habitat genes) 。 多栖息地基因可能来自在多种栖息环境中都生存的物种 , 或者来自基因组之间或者跨越栖息地边界水平转移的移动遗传原件(mobile elements) 。 研究发现 , 大多数基因都是栖息环境特异性的 , 这与微生物倾向于适应环境的特性是一致的;只有5.8%物种水平的单基因簇是多栖息环境基因 , 多栖息环境基因主要富集在抗生素耐药性基因和移动遗传原件 。
研究者们进一步研究了宏基因组中单基因簇的频率 , 发现大多数单基因簇是出现频率低于0.1%的罕见基因 。 单基因簇的频率服从中性(或接近中性)进化假设下的幂律分布(power law) 。 事实上 , 虽然观察到很多变异 , 但大多数变异并不是对环境的适应 , 而是由所谓的“中性进化”驱动:变异只是随机的结果 , 而不是达尔文选择 。
这些发现对于理解抗生素抗性的产生 , 以及未来抗菌药物的研发具有重要的意义 。
多学科交融的国际化研究团队
复旦大学类脑智能科学与技术研究院生物医学人工智能团队聚焦于人工智能与生物医学交叉研究 , 自2018年以来引进一批国内外优秀学者 。 团队拥有计算机、数学、生物、物理等不同学科背景 , 交叉融合 , 国际化氛围浓厚 。 2020年 , 团队获吴文俊人工智能自然科学一等奖 。
作为类脑研究院生物医学人工智能团队的负责人 , 赵兴明介绍 , 前沿科学越来越突破学科界限 , 需要全域视野和全球视野 。 本研究构建了一个全球视域下的微生物基因目录 , 对于理解微生物与人类健康的关系具有重要的作用 。
特别声明:本站内容均来自网友提供或互联网,仅供参考,请勿用于商业和其他非法用途。如果侵犯了您的权益请与我们联系,我们将在24小时内删除。
