文章图片
(3)食管鳞癌和配对正常食管上皮组织之间差异表达的eccDNA鉴定
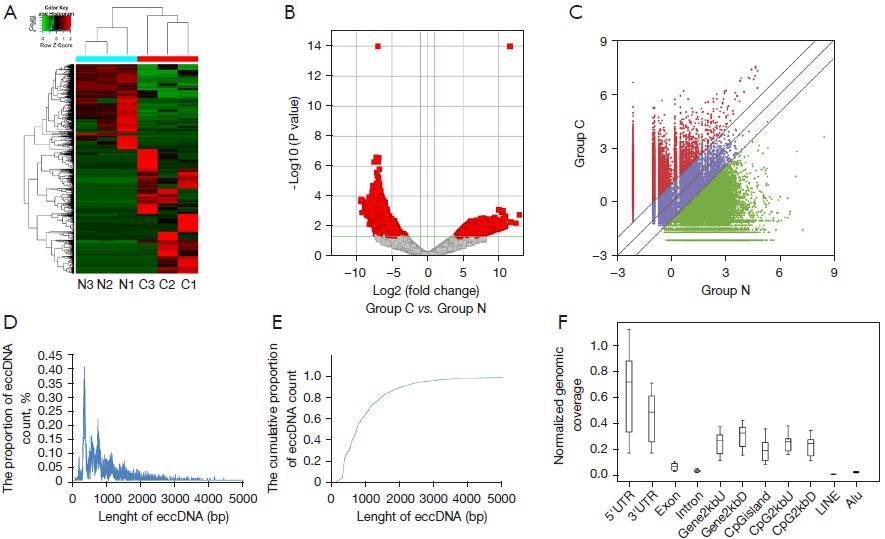
分析了两组样本的eccDNA分布模式后 , 作者接着比较了食管鳞癌和正常食管上皮中eccDNA的表达水平差异 。 共有16031个差异表达的eccDNA(P-value<0.05, |LogFC| >1) , 其中上调10126个 , 下调5905个;且只有一小部分差异eccDNA(候选功能性eccDNA)在食管鳞癌和正常食管样本中均检测到 。 这些候选eccDNA可能是食管鳞癌的潜在生物标志物 , 并可能参与食管鳞癌的发生和发展 。 其长度分布范围为44~395,264 bp , 且它们主要来自5'UTR和3'UTR 。 经过分析发现这些上调的eccDNA来源于3219个基因 , 主要是转录和发育调节因子2(AUTS2)基因 。 下调的eccDNA来自2235个基因 , 其中LSAMP、CUB和CSMD1和BICD1基因产生的eccDNA最多 。
文章图片
(4) 差异表达eccDNA相关基因的GO和KEGG通路分析
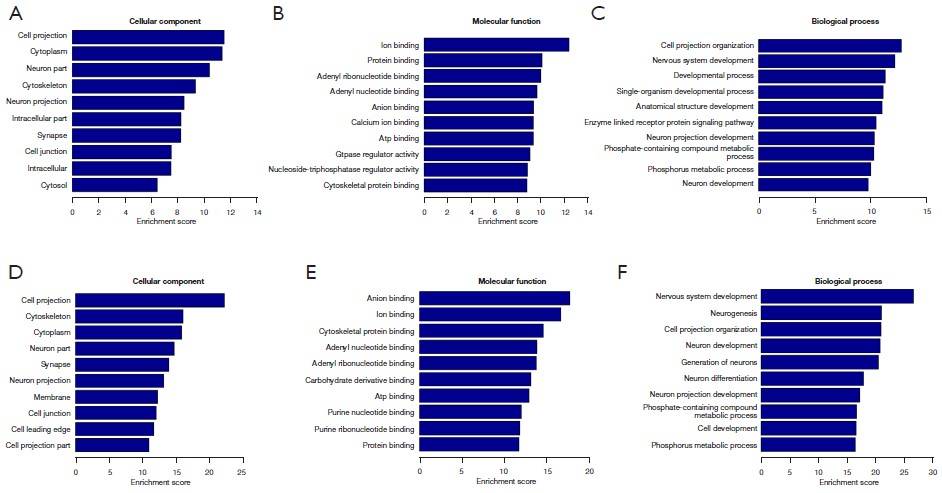
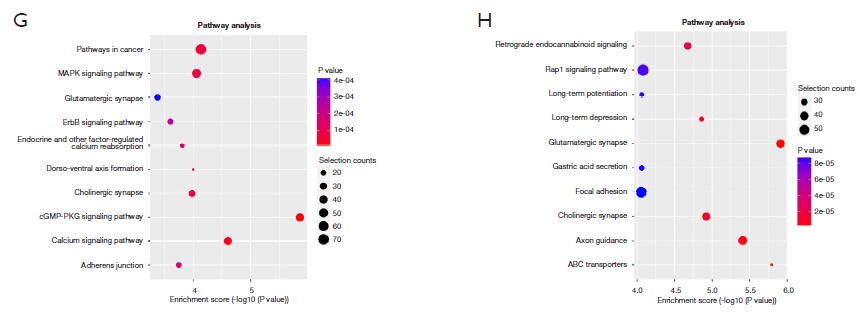
为了探究差异表达eccDNA相关基因的功能 , 进行了GO和KEGG分析 。 发现这些基因主要与神经元、GTPase相关活性和细胞骨架等有关 。 此外 , KEGG通路分析显示 , 差异表达的eccDNA相关基因主要涉及癌症相关通路、促丝分裂原活化蛋白激酶(MAPK)通路、局灶粘附和Rap1通路 。
文章图片
文章图片
(5)食管鳞癌样品中eccDNA的验证
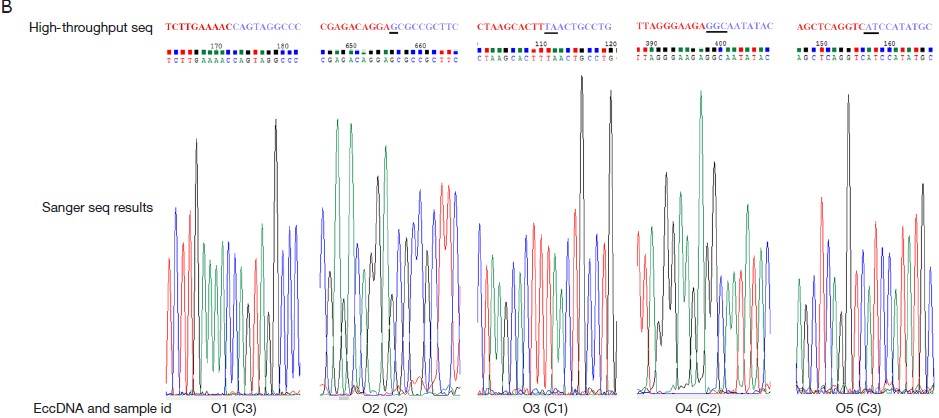
作者接着挑选了部分eccDNA进行PCR验证 , 包括5个上调和下调的指标 。 其中 , 5个上调的eccDNA在至少一种肿瘤样本中强烈表达 , 而在所有正常上皮样本中均为阴性 。 同时 , 5个下调的eccdna在所有肿瘤样本和正常上皮样本中均为阴性 。 为分析其原因 , 将高通量测序数据与PCR结果进行了比较 , 发现只有原始表达值高的eccDNA分子能在PCR中检测到 。
通过TOPO-TA克隆和Sanger测序验证反式剪切位点 。 其中 , 5个上调的eccDNA连接位点在至少一个PCR阳性产物中被验证到 。 当eccDNA的起始位点和末端位点的基因组下游有一个或多个相同的核苷酸时 , 由于高通量测序无法确定连接位点上相同的核苷酸是来自起始位点还是来自末端位点 , 因此高通量测序显示的末端位点将延伸到真正末端位点下游的一个或多个核苷酸 。 综上所述 , 以上验证结果与高通量测序结果一致 。
文章图片
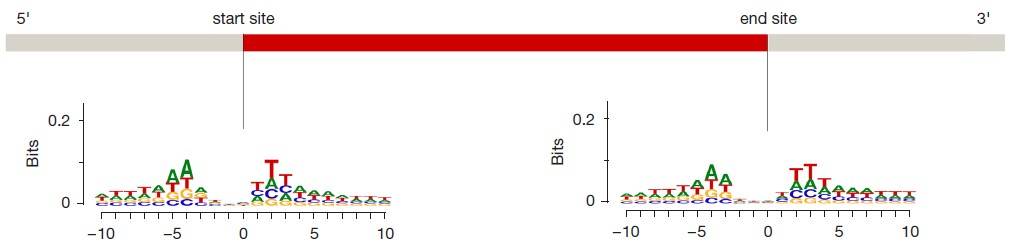
(6)eccDNA成环连接处的motif分析
为探讨eccDNA的产生机制 , 对每个eccDNA起始和结束位置上游10 bp至下游10 bp的核苷酸组成进行了motif分析 。 有趣的是 , 在起始位点和结束位点之间检测到重复的核苷酸模式 , 这种核苷酸模式可能是导致eccDNA形成的原因 。
文章图片
总结:本文通过对食管鳞癌和配对正常组织进行eccDNA测序(circle-seq) , 首次揭示了食管鳞癌的eccDNA表达谱 , 阐明了癌症组和对照组中eccDNA的染色体、基因组以及长度分布情况 。 并对两组进行差异分析 , 得到差异上下调的eccDNA , 通过功能分析发现主要富集在癌症相关通路 , 这些eccDNA可能是食管鳞癌的潜在生物标志物 , 并参与食管鳞癌的发生和发展 。 最后通过qPCR和Sanger测序验证目标eccDNA 。 本研究为我们理解基因组可塑性、eccDNA在食管鳞癌中的作用提供了进一步的见解 , 并可能有助于潜在的临床治疗发展 。
特别声明:本站内容均来自网友提供或互联网,仅供参考,请勿用于商业和其他非法用途。如果侵犯了您的权益请与我们联系,我们将在24小时内删除。
