机器之心报道
编辑:小舟、蛋酱
AlphaFold 虽好 , 但耗时且成本高 , 现在首个用于蛋白质结构预测模型的性能优化方案来了 。蛋白质结构预测一直是结构生物学中的一个重要的研究问题 。 直接从蛋白质的氨基酸序列预测蛋白质的三维结构在许多领域都有广泛的应用 , 包括药物设计、蛋白质设计等 。
预测蛋白质结构的方法主要有两种:实验方法和计算方法 。 实验方法需要以高昂的时间和经济成本获得更准确的蛋白质结构 。 计算方法则能够以低成本高吞吐量预测蛋白质结构 , 因此提高计算方法的预测精度至关重要 。 借助深度神经网络 , AlphaFold 能够周期性地以原子精度预测蛋白质结构 , 但 AlphaFold 模型的训练和推理由于其特殊的性能特点和巨大的内存消耗 , 既耗时又昂贵 。
基于此 , 来自潞晨科技和上海交大的研究者提出了一种蛋白质结构预测模型的高效实现 FastFold 。FastFold 包括一系列基于对 AlphaFold 性能全面分析的 GPU 优化 。 同时 , 通过动态轴并行和对偶异步算子 , FastFold 提高了模型并行扩展的效率 , 超越了现有的模型并行方法 。

文章图片
论文地址:https://arxiv.org/abs/2203.00854
实验结果表明 , FastFold 将整体训练时间从 11 天减少到 67 小时 , 并实现了 7.5 ~ 9.5 倍的长序列推理加速 。 此外 , 研究者还将 FastFold 扩展到 512 个 A100 GPU 的超算集群上 , 聚合峰值性能达到了 6.02PetaFLOPs , 扩展效率达到 90.1% 。
FastFold
不同于一般的 Transformer 模型 , AlphaFold 在 GPU 平台上的计算效率较低 , 主要面临两个挑战:1) 有限的全局批大小限制了使用数据并行性将训练扩展到更多节点 , 更大的批大小会导致准确率更低 。 即使使用 128 个谷歌 TPUv3 训练 AlphaFold 也需要约 11 天; 2) 巨大的内存消耗超出了当前 GPU 的处理能力 。 在推理过程中 , 较长的序列对 GPU 内存的需求要大得多 , 对于 AlphaFold 模型 , 一个长序列的推理时间甚至可以达到几个小时 。
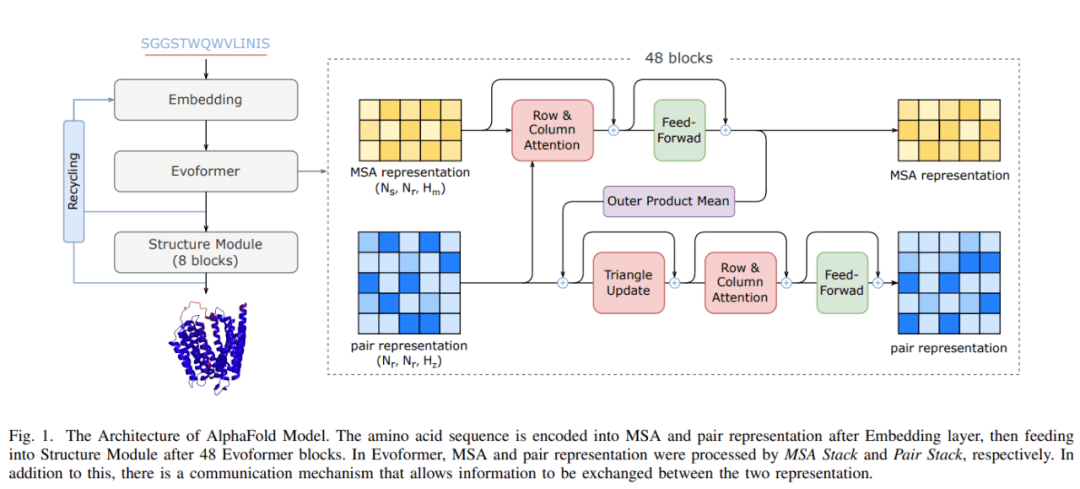
文章图片
AlphaFold 模型架构
作为首个用于蛋白质结构预测模型训练和推理的性能优化工作 , FastFold 成功引入了大型模型训练技术 , 显著降低了 AlphaFold 模型训练和推理的时间和经济成本 。
FastFold 由 Evoformer 的高性能实现、AlphaFold 的主干结构和一种称为动态轴并行(Dynamic Axial Parallelism , DAP)的模型并行新策略组成 。
Evoformer
根据计算和存储访问的特点 , 该研究将算子分为三大类:
1) GEMM 。 这类包括矩阵乘法、批矩阵 - 矩阵相乘和其他密集矩阵计算 。 来自 NVIDIA Tesla GPU 的张量核可以显著加速 GEMM 算子;
2)批递减算子 。 这类包括 LayerNorm、 Softmax 等 , 计算强度低于 GEMM 算子 , 并且更容易出现访问瓶颈 。
3)Element-wise 算子 。 这类包括 element-wise 的相加、相乘、dropout 和激活 , 是计算密集度最低的一类 。
Evoformer 的注意力机制如下图所示
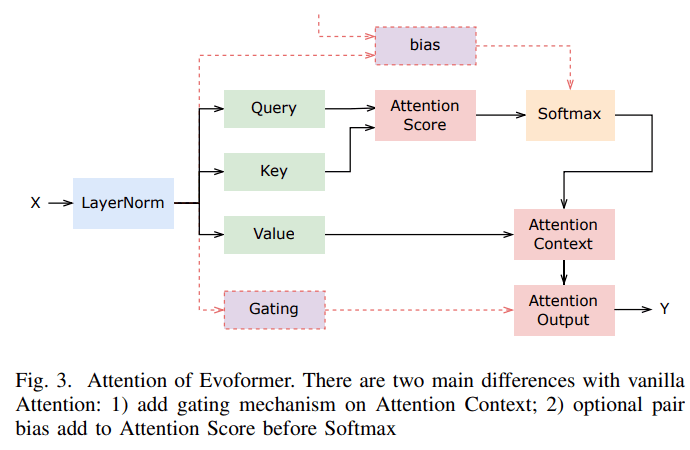
文章图片
值得注意的是 , Evoformer 和原版 Transformer 之间有几个关键区别:
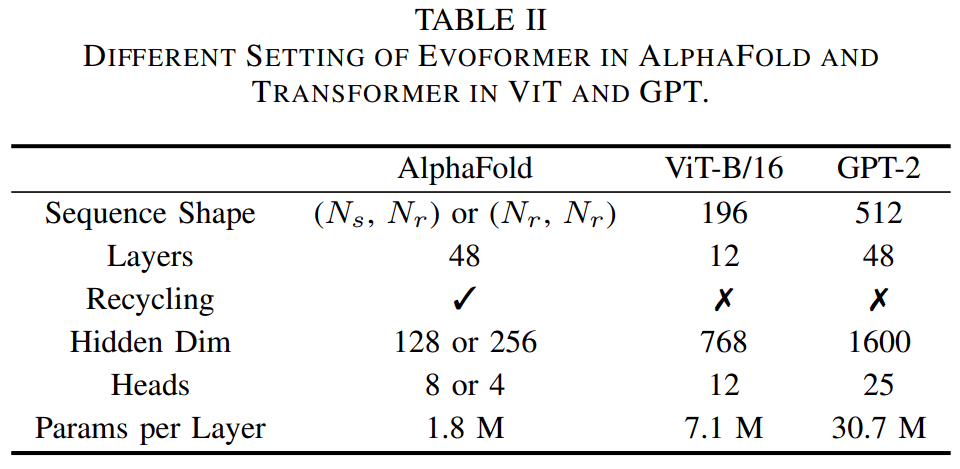
文章图片
该研究分析了 Evoformer 的复杂结构并进行了内核融合 , 并针对 Evoformer 中的特定操作和基于性能特征的特定内核进行了优化 , 例如 Softmax 和 LayerNorm 。 高性能的 Evoformer 实现大大降低了训练和推理的经济成本 。
特别声明:本站内容均来自网友提供或互联网,仅供参考,请勿用于商业和其他非法用途。如果侵犯了您的权益请与我们联系,我们将在24小时内删除。
