与GO富集分析的差异在于GSEA分析不需要指定阈值(p值或FDR)来筛选差异基因,我们可以在没有经验存在的情况下分析我们感兴趣的基因集,而这个基因集不一定是显著差异表达的基因 。GSEA分析可以将那些GO/KEGG富集分信息中容易遗漏掉的差异表达不显著却有着重要生物学意义的基因包含在内 。
下面来看看软件具体操作和结果解读 。
软件下载地址:
http://software.broadinstitute.org/gsea/downloads.jsp
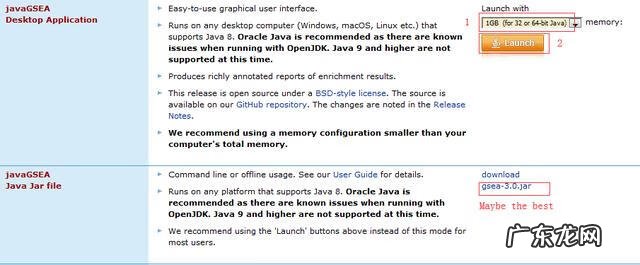
使用官方推荐的第一个软件javaGSEA Desktop Application,根据分析数据的大小和电脑内存多少可以选择下载不同内存版本的软件 。该软件是基于java环境运行的,而且需要联网 。若会出现打不开的现象(小编就是就碰到了),要么是没有安装java,要么是java版本太低了,安装或更新下java就能打开 。也可能是网速太慢,或Java安全性问题,这时选择官网提供的第二个软件javaGSEA Java Jar file,同样依赖java运行,但不需联网,启动快 。
文章插图
软件启动界面如下:

文章插图
所有矩阵的列以tab键分割,不同类型的数据格式和后缀要求见下表 。
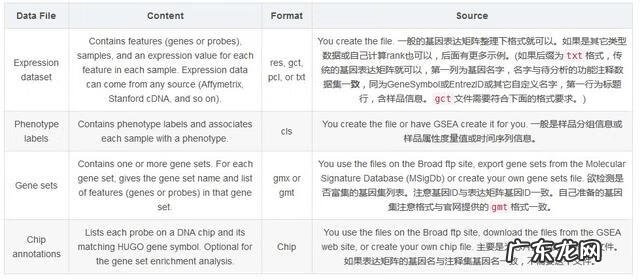
文章插图
Content
Format
Source
Expression dataset
Contains features (genes or probes), samples, and an expression value for each feature in each sample. Expression data can come from any source (Affymetrix, Stanford cDNA, and so on).
res, gct, pcl, or txt
You create the file. 一般的基因表达矩阵整理下格式就可以 。如果是其它类型数据或自己计算rank也可以,后面有更多示例 。(如果后缀为txt格式,传统的基因表达矩阵就可以,第一列为基因名字,名字与待分析的功能注释数据集一致,同为GeneSymbol或EntrezID或其它自定义名字,第一行为标题行,含样品信息 。gct文件需要符合下面的格式要求 。)
Phenotype labels
Contains phenotype labels and associates each sample with a phenotype.
cls
You create the file or have GSEA create it for you. 一般是样品分组信息或样品属性度量值或时间序列信息 。
Gene sets
Contains one or more gene sets. For each gene set, gives the gene set name and list of features (genes or probes) in that gene set.
gmx or gmt
You use the files on the Broad ftp site, export gene sets from the Molecular Signature Database (MSigDb) or create your own gene sets file. 欲检测是否富集的基因集列表 。注意基因ID与表达矩阵基因ID一致 。自己准备的基因集注意格式与官网提供的gmt格式一致 。
Chip annotations
Lists each probe on a DNA chip and its matching HUGO gene symbol. Optional for the gene set enrichment analysis.
Chip
You use the files on the Broad ftp site, download the files from the GSEA web site, or create your own chip file. 主要是为芯片探针设计的转换文件 。如果表达矩阵的基因名与注释集基因名一致,不需要这个文件 。
Data File
Content
Format
Source
Expression dataset
Contains features (genes or probes), samples, and an expression value for each feature in each sample. Expression data can come from any source (Affymetrix, Stanford cDNA, and so on).
res, gct, pcl, or txt
You create the file. 一般的基因表达矩阵整理下格式就可以 。如果是其它类型数据或自己计算rank也可以,后面有更多示例 。(如果后缀为txt格式,传统的基因表达矩阵就可以,第一列为基因名字,名字与待分析的功能注释数据集一致,同为GeneSymbol或EntrezID或其它自定义名字,第一行为标题行,含样品信息 。gct文件需要符合下面的格式要求 。)
- 中国研究生准考证打印 研究生考试准考证打印要求
- 英语四级考试考什么 四级英语总分多少
- 天津会计初级考试 天津会计考试
- 四级考试时间 四级考试报名条件
- 英语口语考试常用对话 英语口语考试对话内容
- 公共英语等级有几级 全国公共英语等级考试
- 会计初级证书报名条件 会计初级职称考试报考条件
- 造价工程师入门手册书籍 造价师考试用书
- 幼儿教师招聘考试试题及答案?
- 广东省公务员考试成绩怎么查询?
特别声明:本站内容均来自网友提供或互联网,仅供参考,请勿用于商业和其他非法用途。如果侵犯了您的权益请与我们联系,我们将在24小时内删除。
