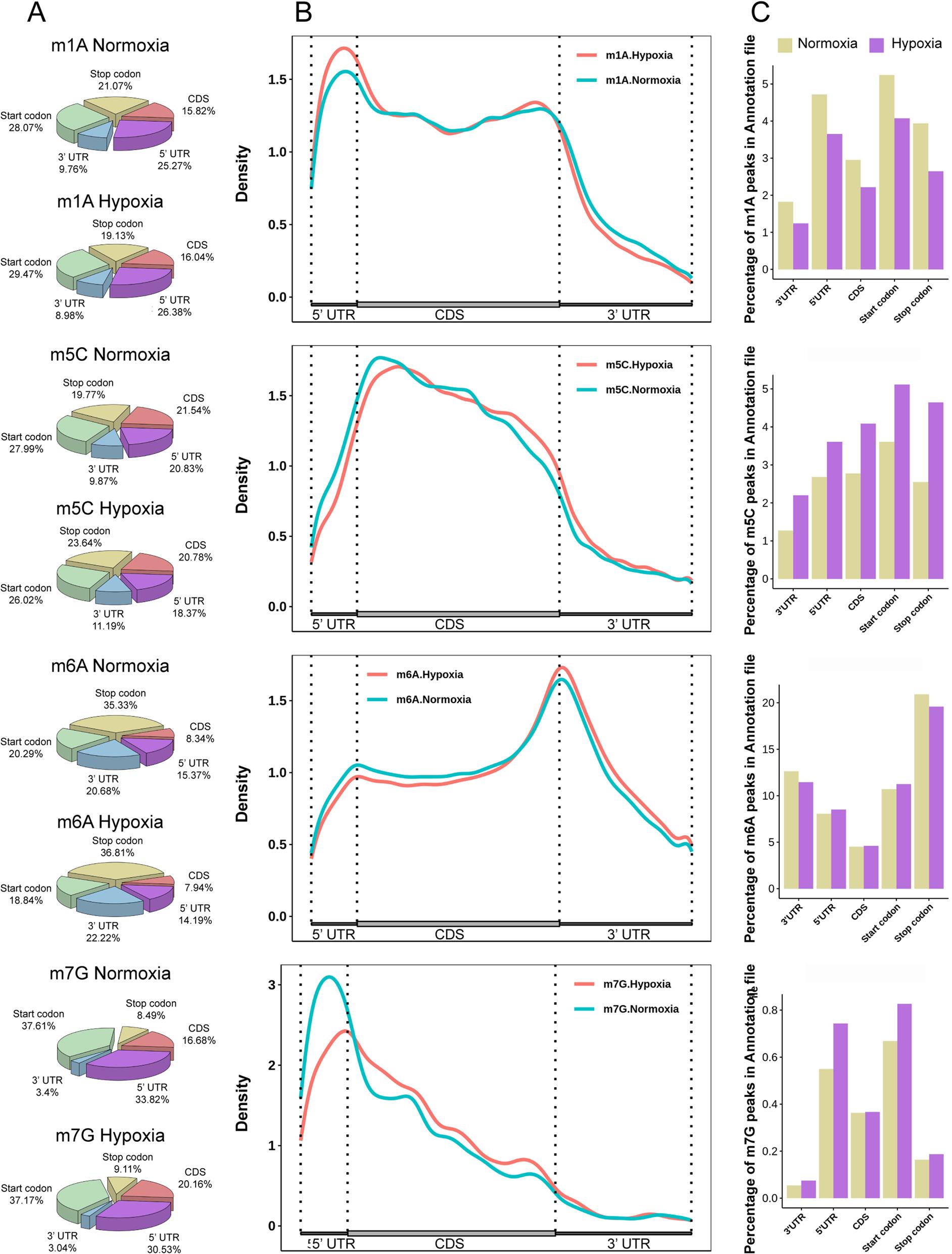
2、m1A, m5C, m6A和m7G在mRNA中的分布特征
为了更好地了解m1A、m6A、m5C和m7G位点在斑马鱼脑组织中的分布 , 分析了其在基因组上的分布特征 。 发现m1A和m7G倾向于富集在5’UTR和起始密码子区域 , 而m5C富集在CDS区域 , m6A富集在终止密码子附近 。 此外 ,m1A丰度在缺氧的CDS区下调 , 而m5C丰度在缺氧的CDS区上调 。 m6A丰度无差异 , 但5’UTR和起始密码子区域中m7G丰度下调 。 以上结果表明 , 在缺氧条件下 , 斑马鱼脑组织的四种甲基化修饰表现出显著差异 。
文章图片
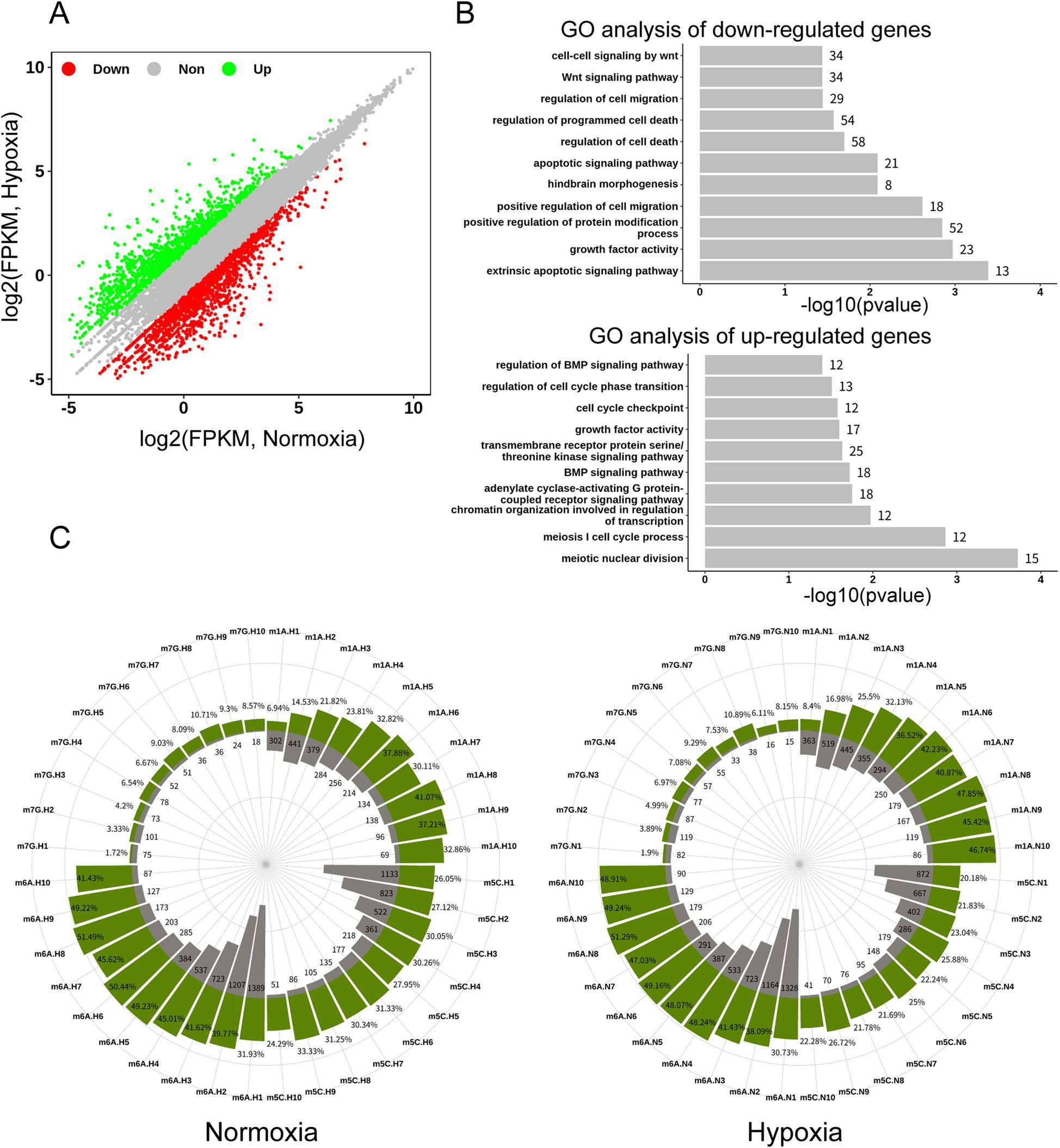
3、差异表达基因的GO功能以及基因表达与RNA甲基化的关联分析
接下来 , 为了探究缺氧处理后RNA表达水平的变化 , 对脑组织样本进行了mRNA-seq 。 共鉴定出3087个差异表达基因 。 通过GO功能分析发现这些差异表达基因与斑马鱼大脑发育相关的后脑形态发生期显著相关 。 此外 , 作者还发现与轴突损伤相关的基因表达发生了改变 , 如Socs3a、Dpysl2b、Neurog1 。 提示脑损伤可能与缺氧诱导有关 。
接下来 , 对RNA甲基化和RNA表达进行了关联分析 。 随着基因表达水平升高 , 这四种修饰的总体水平也逐渐升高 。
文章图片
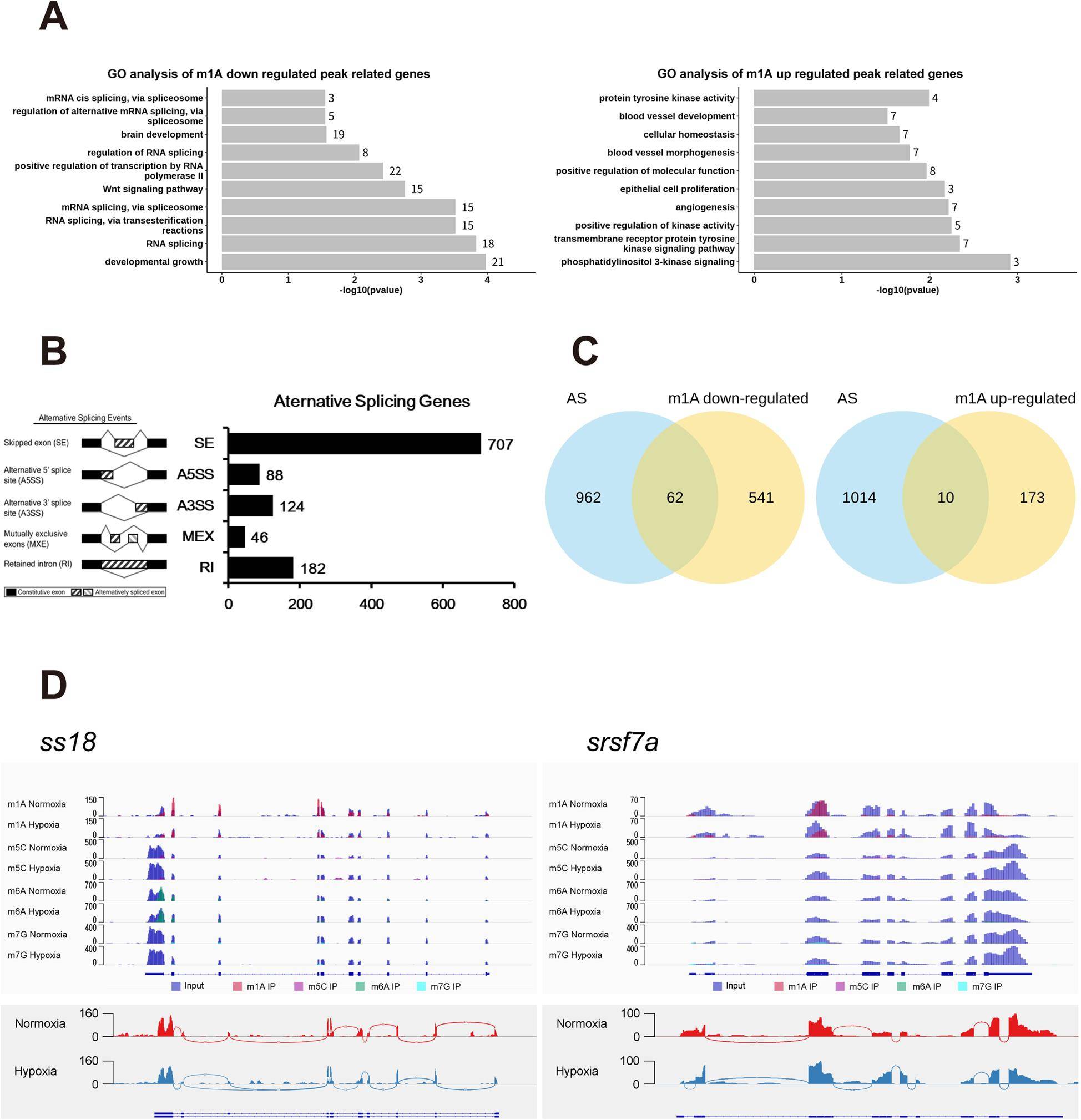
4、斑马鱼脑中高度保守的内部m1A修饰与部分mRNA的剪接有关
在斑马鱼脑组织中以m1A修饰最为丰富 , 根据m1A-MeRIP-seq测序结果对差异甲基化修饰的基因进行了GO富集分析 。 结果显示这些差异基因主要参与激酶活性、可变RNA剪接、Wnt信号通路等 。 一项研究指出 , 在人类基因组中 , 95%包含多个外显子的基因都具有选择性剪接(AS) 。 因此接下来探究了m1A对RNA剪接的影响 , 发现下调的m1A甲基化基因更有可能被AS靶向 。 其中转录激活因子ss18和剪接因子srsf7在缺氧组大脑中m1A修饰的丰度较低 , 这表明m1A修饰可能参与了AS 。 因此作者推测斑马鱼大脑可能通过改变m1A修饰水平来调节相关基因的表达进而应对缺氧诱导反应 。
文章图片
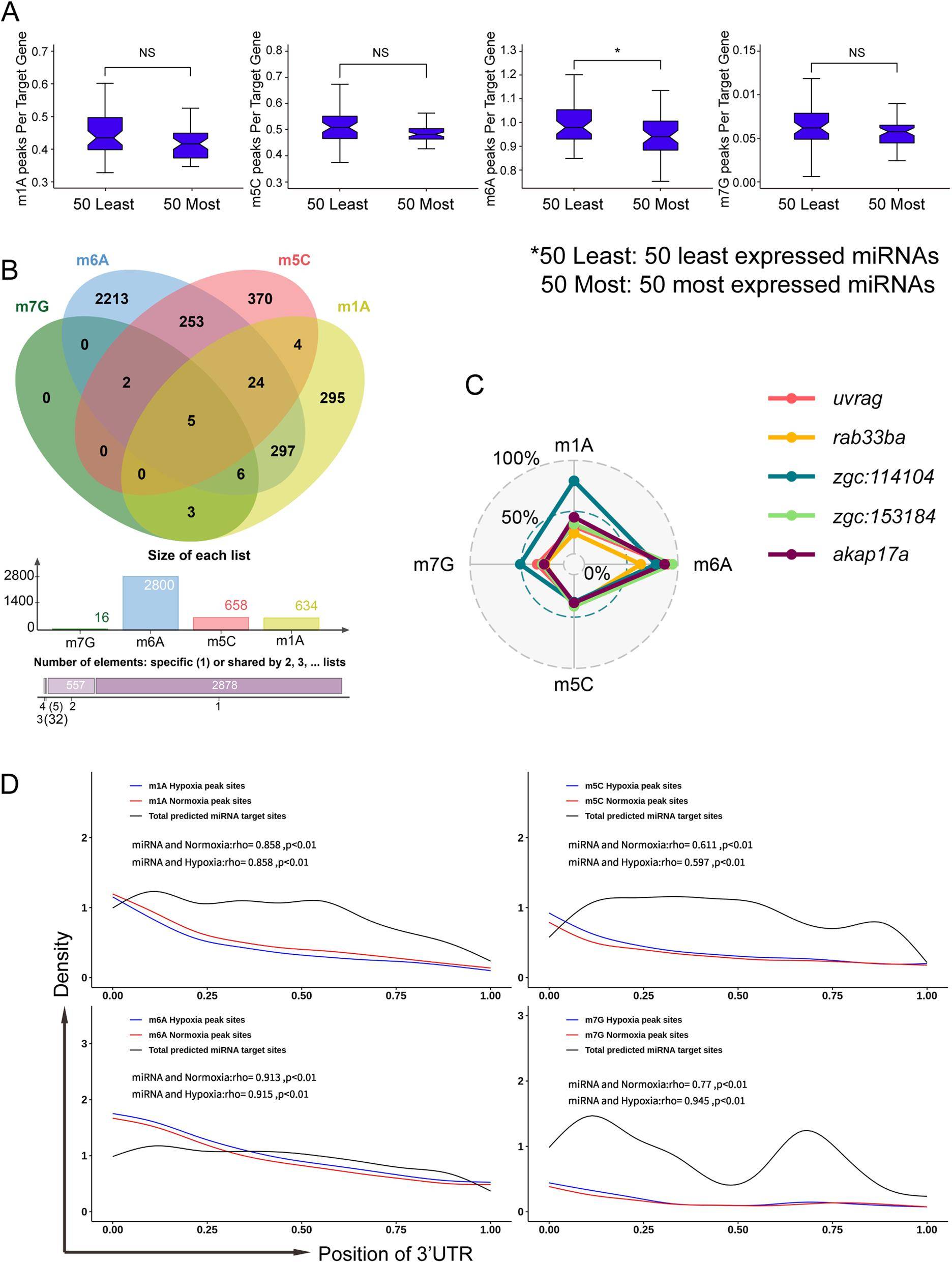
5、3'UTR中m1A、m5C、m6A、m7G与miRNA结合位点的关系
前面的结果显示四种修饰均在3’UTR占一定比例 。 此外,据报道哺乳动物大脑组织有丰富的miRNA,并且可能受m6A调控 。 因此 , 作者接下来分析斑马鱼脑组织中miRNA靶向的转录本是否更容易甲基化 。 结果表明 ,miRNA靶向的5个基因具有m1A、m5C、m6A和m7G甲基化修饰 , 其中m6A的丰度高于其他修饰 。 进一步探究显示miRNA靶选择的偏好与甲基化修饰有关 。 综上所述 , mRNA的四种甲基化修饰可能与3’UTR区域的miRNA结合位点有关 , 其中m6A与miRNA的关系最为显著 。
文章图片
6、斑马鱼脑组织转录本中重复元件的分布及其与m1A、m5C、m6A和m7G RNA甲基化的关系
最近 , 有报道称miRNA来源于内源性逆转录病毒(ERV)和其他转座因子 。 前面的分析显示 , miRNA在斑马鱼脑组织中的分布与RNA甲基化呈正相关 。 进一步分析发现 , 大多数ERV家族重复序列之间没有差异 。 接下来探究了斑马鱼脑转录本中重复元素(REs)的分布 , 发现REs主要富集在3’UTR区 , 与miRNA的分布也呈正相关 。 有研究表明 ,m6A通过转录后修饰调控ERVs的表达 。 结合测序数据 , 发现缺氧处理后 , 大多数REs的m1A和m7G甲基化水平下降 , 尤其是ERV家族 。 这些发现为今后研究脊椎动物脑组织中RNA甲基化修饰对ERV的调控提供了新的视角 。
特别声明:本站内容均来自网友提供或互联网,仅供参考,请勿用于商业和其他非法用途。如果侵犯了您的权益请与我们联系,我们将在24小时内删除。
